澳門科技大學醫學部精準再生醫學研究中心潘星華教授和譚廣亨講座教授聯同南方醫科大學基礎醫學院、珠江醫院和南方醫院的研究人員組成研究團隊在肝細胞癌(HCC)研究領域取得重大突破。
該研究基於HCC小樣本單細胞核測序發現和鑒定了10種不同的肝細胞亞群及其不同的分子圖譜和癌症演進風險,繼而以較大樣本的群體細胞測序進行了初步驗證;並設計了一個新的分位數評分整合方案,有效整合了多個形式各異的獨立隊列大樣本轉錄組數據,結合機器學習,建立了疾病診療模型;還提出了基於分子調控機制的一個肝癌分型新方案,預測出不同亞型的敏感性藥物。這一研究成功揭示了肝癌發生的新機制、新靶點和新分型,為肝癌的早期診斷和治療提供了新的理論依據。這一研究成果近日在國際權威學術期刊 Advanced Science (先進科學雜誌,影響因數14.3,1區)發表“Deciphering the Oncogenic Landscape of Hepatocytes Through Integrated Single-Nucleus and Bulk RNA-Seq of Hepatocellular Carcinoma”。
揭示肝癌發生新機制,破解治療難題
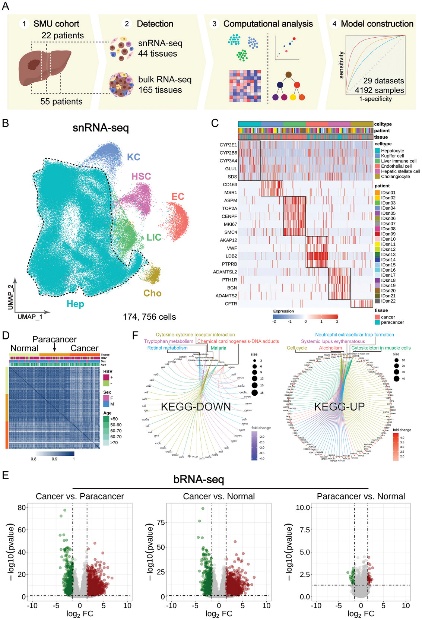
肝癌是全球第六大常見癌症,也是癌症相關死亡的第三大原因,因其早期症狀不明顯,晚期階段治療選擇有限,易復發轉移,預後不理想,臨床診療面臨巨大挑戰。近年基因組學研究的前沿成果和技術為肝癌的防治帶來了新的希望。伴隨這一趨勢,群體細胞轉錄組測序(bRNA-seq)提供了癌症組織的總體轉錄組譜,但往往掩蓋了不同細胞亞群的基因特徵。單細胞轉錄組測序(scRNA-seq)在單細胞的解析度水準解析每個細胞獨立的轉錄組資訊,精準反應細胞水準的功能異質性。但是由於肝細胞較大而且脆弱,以往的全細胞scRNA-seq往往自動富集其他肝微環境細胞, 而剔除了絕大部分肝實質細胞即肝細胞本身,所以迄今對肝細胞本身異質性的理解極為缺乏。單細胞核轉錄組測序(snRNA-seq)能無偏倚地捕獲樣本中的所有細胞核,從而更準確地反映各種組織的天然細胞類型組成,有助於解析肝細胞包括肝癌細胞的內在特徵。該研究結合snRNA-seq和bRNA-seq對77例HCC患者樣本進行了癌症發生和分子調控的綜合分析,發現了關鍵細胞亞群、基因和通路,鑒定了一系列新的診斷治療靶點。研究人員又開發了一種基於分位數的評分方法,有效整合了29個肝癌佇列數據集的4192個樣品的轉錄組資訊,構建了HCC的分位元數分佈模型(QDM),驗證和拓展了實驗結果。在此基礎上,該研究還從分子網路水準重新定義了3類HCC:代謝型HCC、炎症型HCC和基質型HCC,清晰地闡明瞭它們的臨床特徵,並篩選各類HCC的優先候選治療藥物。
影響與重要性
肝癌是全人類特別是亞裔生命健康的重要威脅。研究採用前沿單細胞實驗技術和人工智慧數據分析手段闡明不同肝細胞亞型及其在HCC中的分子特徵和功能作用及其與臨床指標的相關性。利用這些亞型的特徵,該研究基於多中心數據集開發了HCC的高性能診斷模型,並確定了與疾病相關的潛在生物標誌物。該研究提供了迄今爲止最全面的人類肝癌轉錄組資源,並建立了基於肝細胞亞型和調控網路的新型分類系統。這些進展爲進一步探索肝癌發生機制和制定肝癌治療策略提供了扎實的科學依據和獨創性的見解。
關於研究團隊
研究由澳門科技大學和南方醫科大學的研究團隊共同完成,得到了國家自然科學基金、廣東省基礎與應用基礎研究基金,澳門科技大學教師研究基金及澳門科技發展基金等多個項目的支援。澳門科技大學醫學部精準再生醫學研究中心團隊譚廣亨總監、陳嚴助理總監和潘星華教授領銜,本文由潘星華教授擔任首席通訊作者。
有關研究論文,可瀏覽https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202412944





